SIR,SEIR|感染症流行過程の数理モデ
SIRモデル
感染症の短期的な流行過程を決定論的に記述するモデル方程式
$$
\frac{dS(t)}{dt} = -\beta S(t) I(t)
\\
\frac{dI(t)}{dt} = \beta S(t) I(t) – \gamma I(t)
\\
\frac{dR(t)}{dt} = \gamma I(t)
$$
閉鎖的な(人の移動がない)集団を3つの状態に分けます.
- S: 免疫のない感染する可能性のある集団
- I: 感染している人
- R: 感染後に回復した人/死亡した人
パラメータなど
- \(\beta\): 感染率
- \(\gamma\): 回復率や隔離率
- \(\beta I(t)\): 時間tにおける感染力
Pythonによる実装
import numpy as np
import matplotlib.pyplot as plt
from scipy.integrate import odeint
N = 1_000_000
D = 14
R0 = 2
#initial_state
I_0 = 1
R_0 = 0
S_0 = N - I_0 - R_0
#params
gamma = 1/D
beta = R0 * gamma
#time
times =np.arange(0,366, 1)
var_0 = [S_0, I_0, R_0]
def SIR(var, t, N, beta, gamma):
dSdt = - beta*var[0]*var[1]/N
dIdt = beta*var[0]*var[1]/N-gamma*var[1]
dRdt = gamma*var[1]
return [dSdt, dIdt, dRdt]
result = odeint(SIR, var_0, times, args=(beta, gamma))
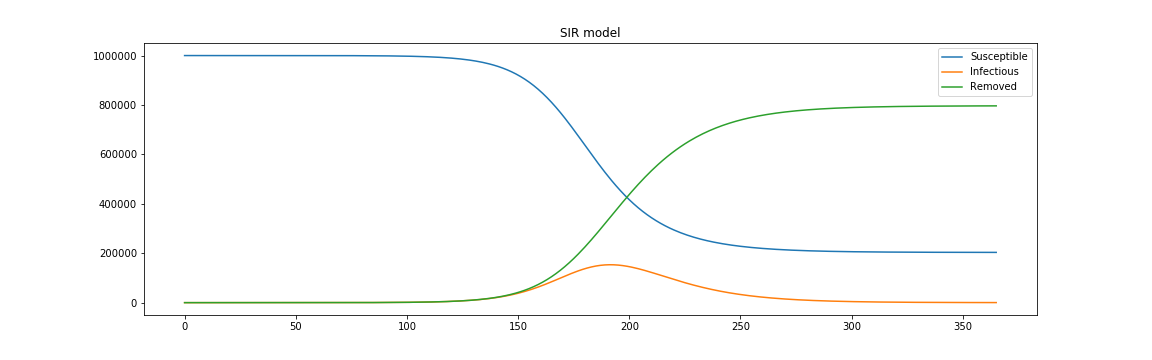
plt.figure(figsize=(16, 5))
plt.plot(times,result)
plt.legend(['Susceptible','Infectious', 'Removed'])
plt.show()
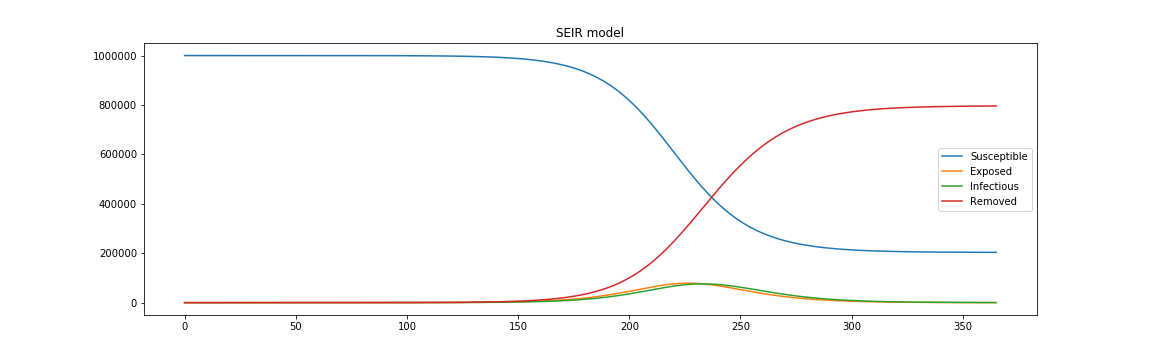
SEIRモデル
$$
\frac{dS(t)}{dt} = -\beta S(t) I(t)
\\
\frac{dE(t)}{dt} = \beta S(t) I(t) – \epsilon E(t)
\\
\frac{dI(t)}{dt} = \epsilon E(t) – \gamma I(t)
\\
\frac{dR(t)}{dt} = \gamma I(t)
$$
閉鎖的な(人の移動がない)集団を4つの状態に分けます.
- S: 免疫のない感染する可能性のある集団
- E: 感染->発症していない人
- I: 感染->発症した人
- R: 感染後に回復した人/死亡した人
- \(\beta\): 感染率
- \(\gamma\): 回復率や隔離率
- \(\epsilon ^{-1}\): 感染待ち時間
- 潜伏期間
Pythonによる実装
import numpy as np
import matplotlib.pyplot as plt
from scipy.integrate import odeint
N = 1_000_000
L = 7
D = 7
R0 = 2
#initial_state
E_0 = 1
I_0 = 0
R_0 = 0
S_0 = N - E_0 - I_0 - R_0
#params
gamma = 1/D
epsilon = 1/L
beta = R0 * epsilon / N
#time
times =np.arange(0,366, 1)
var_0 = [S_0, E_0, I_0, R_0]
def SEIR(var, t, beta, epsilon, gamma):
dSdt = -beta * var[0] * var[2]
dEdt = beta * var[0] * var[2] - epsilon * var[1]
dIdt = epsilon * var[1] - gamma * var[2]
dRdt = gamma * var[2]
return [dSdt, dEdt, dIdt, dRdt]
result = odeint(SEIR, var_0, times, args=(beta, epsilon, gamma))
plt.figure(figsize=(16, 5))
plt.plot(times,result)
plt.legend(['Susceptible', 'Exposed', 'Infectious', 'Removed'])
plt.show()